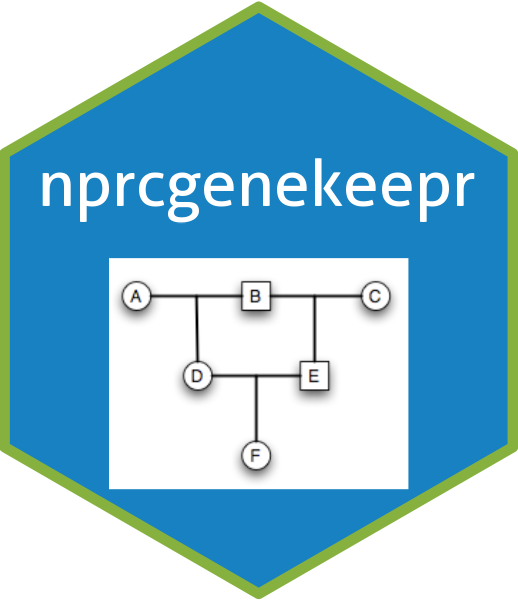
Quick Example of Simulated Kinships with Partial Parentage
nprcgenekeepr: an R Package for the Genetic Management of Colonies
R. Mark Sharp, Ph.D.
8/29/2021
Source:vignettes/simulatedKValues.Rmd
simulatedKValues.RmdIntroduction
This vignette provides a quick example of how to estimate kinship coefficients using simulation. One simulation uses 100 iterations and the other uses the same setup with 1000 simulations to illustrate the type of kinship coefficient estimate variance you can expect with a simple pedigree exhibiting partial parentage for some of the members.
Anticipation of further uses of this kinship estimation method
Retention of founder alleles is a primary driving force behind this package of utilities. This example is intentionally overly simplistic to clearly illustrate this one aspect of the methodology used with realistic pedigrees.
In this example, some of the potential parents are not members of the example pedigree and thus, by definition, have a kinship coeficient of 0.0 with all other pedigree members.
In practice most or all of the potential parents will be members of the same pedigree and will potentially have nonzero kinship coeficients with other pedigree members.
Identification of potential parents
This tutorial assumes knowledge of potential parents and does not present methods for identifying potential parents.
Capabilities to be address with resolution of Issue #281 will provide users the ability to fully automate the identification of potential parents. However, prior to that capability being available, users can use other methods to identify potential parents.
Creation of example pedigree2
The example is based on the following simple pedigree setup. In the pedigree given below, all of the original animals have identifiers that are single letters of the alphabet.:
Only those IDs that have unknown parents are included in the lists. For those IDs with one known parent, the known parent is included in the . See for ID . Note also that potential parents can either come from the pedigree being augmented by simulation or from outside the pedigree. See , , , and for examples of this.
Currently, if you want to provide differential weights for the different potential parents, you will need to do this directly by modifying the number of times each parent is included in the list.
Animals , , and
knitr::opts_chunk$set(echo = TRUE)
library(kableExtra) # nolint: undesirable_function_linter
library(magrittr) # nolint: undesirable_function_linter
library(nprcgenekeepr) # nolint: undesirable_function_linter
library(stringi) # nolint: undesirable_function_linter
ped <- nprcgenekeepr::smallPed
simParent_1 <- list( # nolint: object_name_linter
id = "A", # nolint: object_name_linter
sires = "Q",
dams = c("d1_1", "d1_2", "d1_3", "d1_4")
)
simParent_2 <- list( # nolint: object_name_linter
id = "B", # nolint: object_name_linter
sires = c("s1_1", "s1_2", "s1_3"),
dams = c("d1_1", "d1_2", "d1_3", "d1_4")
)
simParent_3 <- list( # nolint: object_name_linter
id = "E", # nolint: object_name_linter
sires = c("A", "C", "s1_1"),
dams = c("d3_1", "B")
)
simParent_4 <- list( # nolint: object_name_linter
id = "J", # nolint: object_name_linter
sires = c("A", "C", "s1_1"),
dams = c("d3_1", "B")
)
simParent_5 <- list( # nolint: object_name_linter
id = "K", # nolint: object_name_linter
sires = c("A", "C", "s1_1"),
dams = c("d3_1", "d1_2")
)
simParent_6 <- list( # nolint: object_name_linter
id = "N", # nolint: object_name_linter
sires = c("A", "C", "s1_2"),
dams = c("d3_1", "B")
)
allSimParents <- list(
simParent_1, simParent_2, simParent_3,
simParent_4, simParent_5, simParent_6
)
extractKinship <- function(simKinships, id1, id2, simulation) {
ids <- dimnames(simKinships[[simulation]])[[1L]]
simKinships[[simulation]][
seq_along(ids)[ids == id1],
seq_along(ids)[ids == id2]
]
}
extractKValue <- function(kValue, id1, id2, simulation) {
kValue[kValue$id_1 == id1 & kValue$id_2 == id2, paste0("sim_", simulation)]
}Small Example
This is the simulation. I am only printing out rows with kinship values that vary.
Before running these simulations, take time to look at the included function descriptions to see what they are expecting as arguments and what they return.
?createSimKinships
?kinshipMatricesToKValues
?extractKValue
# Only set this seed if you want to get the same simulation results each time.
set.seed(1L)
n <- 10L
simKinships <- createSimKinships(ped, allSimParents, pop = ped$id, n = n)
kValues <- kinshipMatricesToKValues(simKinships)
extractKValue(kValues, id1 = "A", id2 = "F", simulation = 1L:n)## [1] "sim_1" "sim_2" "sim_3" "sim_4" "sim_5" "sim_6" "sim_7" "sim_8"
## [9] "sim_9" "sim_10"
counts <- countKinshipValues(kValues)
counts$kinshipIds[1L:3L]## NULL
counts$kinshipValues[1L:3L]## NULL
counts$kinshipCounts[1L:3L]## NULL
stats_10 <- summarizeKinshipValues(counts)
nrow(stats_10[stats_10$sd > 0.0, ])## [1] 94
kable(stats_10[stats_10$sd > 0.0, ], longtable = TRUE) %>%
kable_styling(
latex_options = c("striped", "repeat_header"),
repeat_header_method = "replace",
repeat_header_text = "\\textit{(continued)}"
)| id_1 | id_2 | min | secondQuartile | mean | median | thirdQuartile | max | sd | |
|---|---|---|---|---|---|---|---|---|---|
| 5 | A | E | 0.00000 | 0.00000 | 0.075000 | 0.000000 | 0.25000 | 0.25000 | 0.1207615 |
| 6 | A | F | 0.12500 | 0.12500 | 0.162500 | 0.125000 | 0.25000 | 0.25000 | 0.0603807 |
| 7 | A | G | 0.12500 | 0.12500 | 0.162500 | 0.125000 | 0.25000 | 0.25000 | 0.0603807 |
| 9 | A | I | 0.25000 | 0.25000 | 0.262500 | 0.250000 | 0.25000 | 0.37500 | 0.0395285 |
| 10 | A | J | 0.00000 | 0.00000 | 0.025000 | 0.000000 | 0.00000 | 0.25000 | 0.0790569 |
| 11 | A | K | 0.00000 | 0.00000 | 0.125000 | 0.125000 | 0.25000 | 0.25000 | 0.1317616 |
| 12 | A | L | 0.12500 | 0.12500 | 0.187500 | 0.187500 | 0.25000 | 0.25000 | 0.0658808 |
| 14 | A | N | 0.00000 | 0.00000 | 0.100000 | 0.000000 | 0.25000 | 0.25000 | 0.1290994 |
| 21 | B | E | 0.00000 | 0.00000 | 0.150000 | 0.250000 | 0.25000 | 0.25000 | 0.1290994 |
| 22 | B | F | 0.12500 | 0.12500 | 0.200000 | 0.250000 | 0.25000 | 0.25000 | 0.0645497 |
| 23 | B | G | 0.12500 | 0.12500 | 0.200000 | 0.250000 | 0.25000 | 0.25000 | 0.0645497 |
| 25 | B | I | 0.00000 | 0.00000 | 0.062500 | 0.062500 | 0.12500 | 0.12500 | 0.0658808 |
| 26 | B | J | 0.00000 | 0.00000 | 0.125000 | 0.125000 | 0.25000 | 0.25000 | 0.1317616 |
| 30 | B | N | 0.00000 | 0.00000 | 0.100000 | 0.000000 | 0.25000 | 0.25000 | 0.1290994 |
| 36 | C | E | 0.00000 | 0.00000 | 0.112500 | 0.125000 | 0.25000 | 0.25000 | 0.1094494 |
| 37 | C | F | 0.12500 | 0.12500 | 0.181250 | 0.187500 | 0.25000 | 0.25000 | 0.0547247 |
| 38 | C | G | 0.12500 | 0.12500 | 0.181250 | 0.187500 | 0.25000 | 0.25000 | 0.0547247 |
| 40 | C | I | 0.12500 | 0.12500 | 0.162500 | 0.156250 | 0.18750 | 0.25000 | 0.0437004 |
| 41 | C | J | 0.00000 | 0.00000 | 0.075000 | 0.062500 | 0.12500 | 0.25000 | 0.0874007 |
| 42 | C | K | 0.00000 | 0.00000 | 0.062500 | 0.062500 | 0.12500 | 0.12500 | 0.0658808 |
| 43 | C | L | 0.25000 | 0.25000 | 0.281250 | 0.281250 | 0.31250 | 0.31250 | 0.0329404 |
| 45 | C | N | 0.00000 | 0.00000 | 0.100000 | 0.125000 | 0.12500 | 0.12500 | 0.0527046 |
| 50 | D | E | 0.00000 | 0.00000 | 0.112500 | 0.125000 | 0.25000 | 0.25000 | 0.1094494 |
| 51 | D | F | 0.25000 | 0.25000 | 0.306250 | 0.312500 | 0.37500 | 0.37500 | 0.0547247 |
| 52 | D | G | 0.25000 | 0.25000 | 0.306250 | 0.312500 | 0.37500 | 0.37500 | 0.0547247 |
| 54 | D | I | 0.12500 | 0.12500 | 0.162500 | 0.156250 | 0.18750 | 0.25000 | 0.0437004 |
| 55 | D | J | 0.00000 | 0.00000 | 0.075000 | 0.062500 | 0.12500 | 0.25000 | 0.0874007 |
| 56 | D | K | 0.00000 | 0.00000 | 0.062500 | 0.062500 | 0.12500 | 0.12500 | 0.0658808 |
| 57 | D | L | 0.12500 | 0.12500 | 0.156250 | 0.156250 | 0.18750 | 0.18750 | 0.0329404 |
| 59 | D | N | 0.00000 | 0.00000 | 0.100000 | 0.125000 | 0.12500 | 0.12500 | 0.0527046 |
| 64 | E | F | 0.25000 | 0.25000 | 0.306250 | 0.312500 | 0.37500 | 0.37500 | 0.0547247 |
| 65 | E | G | 0.25000 | 0.25000 | 0.306250 | 0.312500 | 0.37500 | 0.37500 | 0.0547247 |
| 66 | E | H | 0.00000 | 0.00000 | 0.112500 | 0.125000 | 0.25000 | 0.25000 | 0.1094494 |
| 67 | E | I | 0.00000 | 0.00000 | 0.068750 | 0.031250 | 0.12500 | 0.25000 | 0.0905711 |
| 68 | E | J | 0.00000 | 0.00000 | 0.062500 | 0.000000 | 0.12500 | 0.25000 | 0.0883883 |
| 69 | E | K | 0.00000 | 0.00000 | 0.087500 | 0.125000 | 0.12500 | 0.12500 | 0.0603807 |
| 70 | E | L | 0.00000 | 0.00000 | 0.100000 | 0.093750 | 0.18750 | 0.18750 | 0.0733617 |
| 71 | E | M | 0.00000 | 0.00000 | 0.037500 | 0.000000 | 0.12500 | 0.12500 | 0.0603807 |
| 72 | E | N | 0.00000 | 0.00000 | 0.050000 | 0.000000 | 0.12500 | 0.25000 | 0.0874007 |
| 74 | E | P | 0.00000 | 0.00000 | 0.018750 | 0.000000 | 0.06250 | 0.06250 | 0.0301904 |
| 75 | E | Q | 0.00000 | 0.00000 | 0.037500 | 0.000000 | 0.12500 | 0.12500 | 0.0603807 |
| 76 | F | F | 0.50000 | 0.50000 | 0.556250 | 0.562500 | 0.62500 | 0.62500 | 0.0547247 |
| 77 | F | G | 0.25000 | 0.25000 | 0.306250 | 0.312500 | 0.37500 | 0.37500 | 0.0547247 |
| 78 | F | H | 0.12500 | 0.12500 | 0.181250 | 0.187500 | 0.25000 | 0.25000 | 0.0547247 |
| 79 | F | I | 0.06250 | 0.06250 | 0.115625 | 0.093750 | 0.12500 | 0.25000 | 0.0608283 |
| 80 | F | J | 0.00000 | 0.00000 | 0.068750 | 0.062500 | 0.12500 | 0.25000 | 0.0804177 |
| 81 | F | K | 0.00000 | 0.00000 | 0.075000 | 0.062500 | 0.12500 | 0.12500 | 0.0395285 |
| 82 | F | L | 0.06250 | 0.06250 | 0.128125 | 0.125000 | 0.18750 | 0.18750 | 0.0452856 |
| 83 | F | M | 0.06250 | 0.06250 | 0.081250 | 0.062500 | 0.12500 | 0.12500 | 0.0301904 |
| 84 | F | N | 0.00000 | 0.00000 | 0.075000 | 0.062500 | 0.12500 | 0.18750 | 0.0574335 |
| 86 | F | P | 0.03125 | 0.03125 | 0.040625 | 0.031250 | 0.06250 | 0.06250 | 0.0150952 |
| 87 | F | Q | 0.06250 | 0.06250 | 0.081250 | 0.062500 | 0.12500 | 0.12500 | 0.0301904 |
| 88 | G | G | 0.50000 | 0.50000 | 0.556250 | 0.562500 | 0.62500 | 0.62500 | 0.0547247 |
| 89 | G | H | 0.12500 | 0.12500 | 0.181250 | 0.187500 | 0.25000 | 0.25000 | 0.0547247 |
| 90 | G | I | 0.06250 | 0.06250 | 0.115625 | 0.093750 | 0.12500 | 0.25000 | 0.0608283 |
| 91 | G | J | 0.00000 | 0.00000 | 0.068750 | 0.062500 | 0.12500 | 0.25000 | 0.0804177 |
| 92 | G | K | 0.00000 | 0.00000 | 0.075000 | 0.062500 | 0.12500 | 0.12500 | 0.0395285 |
| 93 | G | L | 0.06250 | 0.06250 | 0.128125 | 0.125000 | 0.18750 | 0.18750 | 0.0452856 |
| 94 | G | M | 0.06250 | 0.06250 | 0.081250 | 0.062500 | 0.12500 | 0.12500 | 0.0301904 |
| 95 | G | N | 0.00000 | 0.00000 | 0.075000 | 0.062500 | 0.12500 | 0.18750 | 0.0574335 |
| 97 | G | P | 0.03125 | 0.03125 | 0.040625 | 0.031250 | 0.06250 | 0.06250 | 0.0150952 |
| 98 | G | Q | 0.06250 | 0.06250 | 0.081250 | 0.062500 | 0.12500 | 0.12500 | 0.0301904 |
| 100 | H | I | 0.12500 | 0.12500 | 0.162500 | 0.156250 | 0.18750 | 0.25000 | 0.0437004 |
| 101 | H | J | 0.00000 | 0.00000 | 0.075000 | 0.062500 | 0.12500 | 0.25000 | 0.0874007 |
| 102 | H | K | 0.00000 | 0.00000 | 0.062500 | 0.062500 | 0.12500 | 0.12500 | 0.0658808 |
| 103 | H | L | 0.12500 | 0.12500 | 0.156250 | 0.156250 | 0.18750 | 0.18750 | 0.0329404 |
| 105 | H | N | 0.00000 | 0.00000 | 0.100000 | 0.125000 | 0.12500 | 0.12500 | 0.0527046 |
| 109 | I | I | 0.50000 | 0.50000 | 0.512500 | 0.500000 | 0.50000 | 0.62500 | 0.0395285 |
| 110 | I | J | 0.25000 | 0.25000 | 0.262500 | 0.250000 | 0.25000 | 0.37500 | 0.0395285 |
| 111 | I | K | 0.00000 | 0.00000 | 0.075000 | 0.093750 | 0.12500 | 0.18750 | 0.0709558 |
| 112 | I | L | 0.06250 | 0.06250 | 0.118750 | 0.125000 | 0.15625 | 0.21875 | 0.0506040 |
| 113 | I | M | 0.12500 | 0.12500 | 0.131250 | 0.125000 | 0.12500 | 0.18750 | 0.0197642 |
| 114 | I | N | 0.00000 | 0.00000 | 0.068750 | 0.062500 | 0.12500 | 0.18750 | 0.0687816 |
| 116 | I | P | 0.06250 | 0.06250 | 0.065625 | 0.062500 | 0.06250 | 0.09375 | 0.0098821 |
| 117 | I | Q | 0.12500 | 0.12500 | 0.131250 | 0.125000 | 0.12500 | 0.18750 | 0.0197642 |
| 119 | J | K | 0.00000 | 0.00000 | 0.025000 | 0.000000 | 0.00000 | 0.12500 | 0.0527046 |
| 120 | J | L | 0.00000 | 0.00000 | 0.050000 | 0.031250 | 0.06250 | 0.18750 | 0.0645497 |
| 121 | J | M | 0.00000 | 0.00000 | 0.012500 | 0.000000 | 0.00000 | 0.12500 | 0.0395285 |
| 122 | J | N | 0.00000 | 0.00000 | 0.037500 | 0.000000 | 0.12500 | 0.12500 | 0.0603807 |
| 124 | J | P | 0.00000 | 0.00000 | 0.006250 | 0.000000 | 0.00000 | 0.06250 | 0.0197642 |
| 125 | J | Q | 0.00000 | 0.00000 | 0.012500 | 0.000000 | 0.00000 | 0.12500 | 0.0395285 |
| 127 | K | L | 0.25000 | 0.25000 | 0.281250 | 0.281250 | 0.31250 | 0.31250 | 0.0329404 |
| 128 | K | M | 0.00000 | 0.00000 | 0.062500 | 0.062500 | 0.12500 | 0.12500 | 0.0658808 |
| 129 | K | N | 0.00000 | 0.00000 | 0.050000 | 0.000000 | 0.12500 | 0.12500 | 0.0645497 |
| 131 | K | P | 0.00000 | 0.00000 | 0.031250 | 0.031250 | 0.06250 | 0.06250 | 0.0329404 |
| 132 | K | Q | 0.00000 | 0.00000 | 0.062500 | 0.062500 | 0.12500 | 0.12500 | 0.0658808 |
| 133 | L | L | 0.50000 | 0.50000 | 0.531250 | 0.531250 | 0.56250 | 0.56250 | 0.0329404 |
| 134 | L | M | 0.06250 | 0.06250 | 0.093750 | 0.093750 | 0.12500 | 0.12500 | 0.0329404 |
| 135 | L | N | 0.00000 | 0.00000 | 0.075000 | 0.062500 | 0.12500 | 0.12500 | 0.0493007 |
| 137 | L | P | 0.03125 | 0.03125 | 0.046875 | 0.046875 | 0.06250 | 0.06250 | 0.0164702 |
| 138 | L | Q | 0.06250 | 0.06250 | 0.093750 | 0.093750 | 0.12500 | 0.12500 | 0.0329404 |
| 140 | M | N | 0.00000 | 0.00000 | 0.050000 | 0.000000 | 0.12500 | 0.12500 | 0.0645497 |
| 146 | N | P | 0.00000 | 0.00000 | 0.025000 | 0.000000 | 0.06250 | 0.06250 | 0.0322749 |
| 147 | N | Q | 0.00000 | 0.00000 | 0.050000 | 0.000000 | 0.12500 | 0.12500 | 0.0645497 |
A larger simulation
set.seed(1L)
n <- 100L
simKinships <- createSimKinships(ped, allSimParents, pop = ped$id, n = n)
kValues <- kinshipMatricesToKValues(simKinships)
extractKValue(kValues, id1 = "A", id2 = "F", simulation = 1L:10L)## [1] "sim_1" "sim_2" "sim_3" "sim_4" "sim_5" "sim_6" "sim_7" "sim_8"
## [9] "sim_9" "sim_10"
counts <- countKinshipValues(kValues)
counts$kinshipIds[1L:3L]## NULL
counts$kinshipValues[1L:3L]## NULL
counts$kinshipCounts[1L:3L]## NULL
stats_100 <- summarizeKinshipValues(counts)
nrow(stats_100[stats_100$sd > 0.0, ])## [1] 94
kable(stats_100[stats_100$sd > 0.0, ], longtable = TRUE) %>%
kable_styling(
latex_options = c("striped", "repeat_header"),
repeat_header_method = "replace",
repeat_header_text = "\\textit{(continued)}"
)| id_1 | id_2 | min | secondQuartile | mean | median | thirdQuartile | max | sd | |
|---|---|---|---|---|---|---|---|---|---|
| 5 | A | E | 0.00000 | 0.00000 | 0.0725000 | 0.000000 | 0.25000 | 0.25000 | 0.1140120 |
| 6 | A | F | 0.12500 | 0.12500 | 0.1612500 | 0.125000 | 0.25000 | 0.25000 | 0.0570060 |
| 7 | A | G | 0.12500 | 0.12500 | 0.1612500 | 0.125000 | 0.25000 | 0.25000 | 0.0570060 |
| 9 | A | I | 0.25000 | 0.25000 | 0.2825000 | 0.250000 | 0.37500 | 0.37500 | 0.0551055 |
| 10 | A | J | 0.00000 | 0.00000 | 0.0650000 | 0.000000 | 0.25000 | 0.25000 | 0.1102110 |
| 11 | A | K | 0.00000 | 0.00000 | 0.1000000 | 0.000000 | 0.25000 | 0.25000 | 0.1230915 |
| 12 | A | L | 0.12500 | 0.12500 | 0.1750000 | 0.125000 | 0.25000 | 0.25000 | 0.0615457 |
| 14 | A | N | 0.00000 | 0.00000 | 0.0950000 | 0.000000 | 0.25000 | 0.25000 | 0.1219579 |
| 21 | B | E | 0.00000 | 0.00000 | 0.1225000 | 0.000000 | 0.25000 | 0.25000 | 0.1256046 |
| 22 | B | F | 0.12500 | 0.12500 | 0.1862500 | 0.125000 | 0.25000 | 0.25000 | 0.0628023 |
| 23 | B | G | 0.12500 | 0.12500 | 0.1862500 | 0.125000 | 0.25000 | 0.25000 | 0.0628023 |
| 25 | B | I | 0.00000 | 0.00000 | 0.0512500 | 0.000000 | 0.12500 | 0.12500 | 0.0617889 |
| 26 | B | J | 0.00000 | 0.00000 | 0.1025000 | 0.000000 | 0.25000 | 0.25000 | 0.1235778 |
| 30 | B | N | 0.00000 | 0.00000 | 0.1325000 | 0.250000 | 0.25000 | 0.25000 | 0.1254034 |
| 36 | C | E | 0.00000 | 0.00000 | 0.0975000 | 0.125000 | 0.12500 | 0.25000 | 0.0862739 |
| 37 | C | F | 0.12500 | 0.12500 | 0.1737500 | 0.187500 | 0.18750 | 0.25000 | 0.0431369 |
| 38 | C | G | 0.12500 | 0.12500 | 0.1737500 | 0.187500 | 0.18750 | 0.25000 | 0.0431369 |
| 40 | C | I | 0.12500 | 0.12500 | 0.1668750 | 0.187500 | 0.18750 | 0.25000 | 0.0377465 |
| 41 | C | J | 0.00000 | 0.00000 | 0.0837500 | 0.125000 | 0.12500 | 0.25000 | 0.0754929 |
| 42 | C | K | 0.00000 | 0.00000 | 0.0500000 | 0.000000 | 0.12500 | 0.12500 | 0.0615457 |
| 43 | C | L | 0.25000 | 0.25000 | 0.2750000 | 0.250000 | 0.31250 | 0.31250 | 0.0307729 |
| 45 | C | N | 0.00000 | 0.00000 | 0.1137500 | 0.125000 | 0.12500 | 0.25000 | 0.0890023 |
| 50 | D | E | 0.00000 | 0.00000 | 0.0975000 | 0.125000 | 0.12500 | 0.25000 | 0.0862739 |
| 51 | D | F | 0.25000 | 0.25000 | 0.2987500 | 0.312500 | 0.31250 | 0.37500 | 0.0431369 |
| 52 | D | G | 0.25000 | 0.25000 | 0.2987500 | 0.312500 | 0.31250 | 0.37500 | 0.0431369 |
| 54 | D | I | 0.12500 | 0.12500 | 0.1668750 | 0.187500 | 0.18750 | 0.25000 | 0.0377465 |
| 55 | D | J | 0.00000 | 0.00000 | 0.0837500 | 0.125000 | 0.12500 | 0.25000 | 0.0754929 |
| 56 | D | K | 0.00000 | 0.00000 | 0.0500000 | 0.000000 | 0.12500 | 0.12500 | 0.0615457 |
| 57 | D | L | 0.12500 | 0.12500 | 0.1500000 | 0.125000 | 0.18750 | 0.18750 | 0.0307729 |
| 59 | D | N | 0.00000 | 0.00000 | 0.1137500 | 0.125000 | 0.12500 | 0.25000 | 0.0890023 |
| 64 | E | F | 0.25000 | 0.25000 | 0.2987500 | 0.312500 | 0.31250 | 0.37500 | 0.0431369 |
| 65 | E | G | 0.25000 | 0.25000 | 0.2987500 | 0.312500 | 0.31250 | 0.37500 | 0.0431369 |
| 66 | E | H | 0.00000 | 0.00000 | 0.0975000 | 0.125000 | 0.12500 | 0.25000 | 0.0862739 |
| 67 | E | I | 0.00000 | 0.00000 | 0.0643750 | 0.062500 | 0.12500 | 0.25000 | 0.0702039 |
| 68 | E | J | 0.00000 | 0.00000 | 0.0562500 | 0.000000 | 0.12500 | 0.25000 | 0.0782039 |
| 69 | E | K | 0.00000 | 0.00000 | 0.0312500 | 0.000000 | 0.06250 | 0.12500 | 0.0543993 |
| 70 | E | L | 0.00000 | 0.00000 | 0.0643750 | 0.062500 | 0.12500 | 0.18750 | 0.0578818 |
| 71 | E | M | 0.00000 | 0.00000 | 0.0362500 | 0.000000 | 0.12500 | 0.12500 | 0.0570060 |
| 72 | E | N | 0.00000 | 0.00000 | 0.0587500 | 0.000000 | 0.12500 | 0.25000 | 0.0742279 |
| 74 | E | P | 0.00000 | 0.00000 | 0.0181250 | 0.000000 | 0.06250 | 0.06250 | 0.0285030 |
| 75 | E | Q | 0.00000 | 0.00000 | 0.0362500 | 0.000000 | 0.12500 | 0.12500 | 0.0570060 |
| 76 | F | F | 0.50000 | 0.50000 | 0.5487500 | 0.562500 | 0.56250 | 0.62500 | 0.0431369 |
| 77 | F | G | 0.25000 | 0.25000 | 0.2987500 | 0.312500 | 0.31250 | 0.37500 | 0.0431369 |
| 78 | F | H | 0.12500 | 0.12500 | 0.1737500 | 0.187500 | 0.18750 | 0.25000 | 0.0431369 |
| 79 | F | I | 0.06250 | 0.06250 | 0.1156250 | 0.093750 | 0.12500 | 0.25000 | 0.0429490 |
| 80 | F | J | 0.00000 | 0.00000 | 0.0700000 | 0.062500 | 0.12500 | 0.25000 | 0.0623610 |
| 81 | F | K | 0.00000 | 0.00000 | 0.0406250 | 0.062500 | 0.06250 | 0.12500 | 0.0438581 |
| 82 | F | L | 0.06250 | 0.06250 | 0.1071875 | 0.093750 | 0.12500 | 0.18750 | 0.0341909 |
| 83 | F | M | 0.06250 | 0.06250 | 0.0806250 | 0.062500 | 0.12500 | 0.12500 | 0.0285030 |
| 84 | F | N | 0.00000 | 0.00000 | 0.0862500 | 0.062500 | 0.12500 | 0.25000 | 0.0681071 |
| 86 | F | P | 0.03125 | 0.03125 | 0.0403125 | 0.031250 | 0.06250 | 0.06250 | 0.0142515 |
| 87 | F | Q | 0.06250 | 0.06250 | 0.0806250 | 0.062500 | 0.12500 | 0.12500 | 0.0285030 |
| 88 | G | G | 0.50000 | 0.50000 | 0.5487500 | 0.562500 | 0.56250 | 0.62500 | 0.0431369 |
| 89 | G | H | 0.12500 | 0.12500 | 0.1737500 | 0.187500 | 0.18750 | 0.25000 | 0.0431369 |
| 90 | G | I | 0.06250 | 0.06250 | 0.1156250 | 0.093750 | 0.12500 | 0.25000 | 0.0429490 |
| 91 | G | J | 0.00000 | 0.00000 | 0.0700000 | 0.062500 | 0.12500 | 0.25000 | 0.0623610 |
| 92 | G | K | 0.00000 | 0.00000 | 0.0406250 | 0.062500 | 0.06250 | 0.12500 | 0.0438581 |
| 93 | G | L | 0.06250 | 0.06250 | 0.1071875 | 0.093750 | 0.12500 | 0.18750 | 0.0341909 |
| 94 | G | M | 0.06250 | 0.06250 | 0.0806250 | 0.062500 | 0.12500 | 0.12500 | 0.0285030 |
| 95 | G | N | 0.00000 | 0.00000 | 0.0862500 | 0.062500 | 0.12500 | 0.25000 | 0.0681071 |
| 97 | G | P | 0.03125 | 0.03125 | 0.0403125 | 0.031250 | 0.06250 | 0.06250 | 0.0142515 |
| 98 | G | Q | 0.06250 | 0.06250 | 0.0806250 | 0.062500 | 0.12500 | 0.12500 | 0.0285030 |
| 100 | H | I | 0.12500 | 0.12500 | 0.1668750 | 0.187500 | 0.18750 | 0.25000 | 0.0377465 |
| 101 | H | J | 0.00000 | 0.00000 | 0.0837500 | 0.125000 | 0.12500 | 0.25000 | 0.0754929 |
| 102 | H | K | 0.00000 | 0.00000 | 0.0500000 | 0.000000 | 0.12500 | 0.12500 | 0.0615457 |
| 103 | H | L | 0.12500 | 0.12500 | 0.1500000 | 0.125000 | 0.18750 | 0.18750 | 0.0307729 |
| 105 | H | N | 0.00000 | 0.00000 | 0.1137500 | 0.125000 | 0.12500 | 0.25000 | 0.0890023 |
| 109 | I | I | 0.50000 | 0.50000 | 0.5325000 | 0.500000 | 0.62500 | 0.62500 | 0.0551055 |
| 110 | I | J | 0.25000 | 0.25000 | 0.2825000 | 0.250000 | 0.37500 | 0.37500 | 0.0551055 |
| 111 | I | K | 0.00000 | 0.00000 | 0.0593750 | 0.000000 | 0.12500 | 0.18750 | 0.0655054 |
| 112 | I | L | 0.06250 | 0.06250 | 0.1131250 | 0.109375 | 0.12500 | 0.21875 | 0.0381520 |
| 113 | I | M | 0.12500 | 0.12500 | 0.1412500 | 0.125000 | 0.18750 | 0.18750 | 0.0275528 |
| 114 | I | N | 0.00000 | 0.00000 | 0.0756250 | 0.062500 | 0.12500 | 0.25000 | 0.0760527 |
| 116 | I | P | 0.06250 | 0.06250 | 0.0706250 | 0.062500 | 0.09375 | 0.09375 | 0.0137764 |
| 117 | I | Q | 0.12500 | 0.12500 | 0.1412500 | 0.125000 | 0.18750 | 0.18750 | 0.0275528 |
| 119 | J | K | 0.00000 | 0.00000 | 0.0187500 | 0.000000 | 0.00000 | 0.12500 | 0.0448588 |
| 120 | J | L | 0.00000 | 0.00000 | 0.0512500 | 0.062500 | 0.06250 | 0.18750 | 0.0464830 |
| 121 | J | M | 0.00000 | 0.00000 | 0.0325000 | 0.000000 | 0.12500 | 0.12500 | 0.0551055 |
| 122 | J | N | 0.00000 | 0.00000 | 0.0562500 | 0.000000 | 0.12500 | 0.25000 | 0.0673614 |
| 124 | J | P | 0.00000 | 0.00000 | 0.0162500 | 0.000000 | 0.06250 | 0.06250 | 0.0275528 |
| 125 | J | Q | 0.00000 | 0.00000 | 0.0325000 | 0.000000 | 0.12500 | 0.12500 | 0.0551055 |
| 127 | K | L | 0.25000 | 0.25000 | 0.2750000 | 0.250000 | 0.31250 | 0.31250 | 0.0307729 |
| 128 | K | M | 0.00000 | 0.00000 | 0.0500000 | 0.000000 | 0.12500 | 0.12500 | 0.0615457 |
| 129 | K | N | 0.00000 | 0.00000 | 0.0337500 | 0.000000 | 0.12500 | 0.12500 | 0.0557745 |
| 131 | K | P | 0.00000 | 0.00000 | 0.0250000 | 0.000000 | 0.06250 | 0.06250 | 0.0307729 |
| 132 | K | Q | 0.00000 | 0.00000 | 0.0500000 | 0.000000 | 0.12500 | 0.12500 | 0.0615457 |
| 133 | L | L | 0.50000 | 0.50000 | 0.5250000 | 0.500000 | 0.56250 | 0.56250 | 0.0307729 |
| 134 | L | M | 0.06250 | 0.06250 | 0.0875000 | 0.062500 | 0.12500 | 0.12500 | 0.0307729 |
| 135 | L | N | 0.00000 | 0.00000 | 0.0737500 | 0.062500 | 0.12500 | 0.18750 | 0.0585089 |
| 137 | L | P | 0.03125 | 0.03125 | 0.0437500 | 0.031250 | 0.06250 | 0.06250 | 0.0153864 |
| 138 | L | Q | 0.06250 | 0.06250 | 0.0875000 | 0.062500 | 0.12500 | 0.12500 | 0.0307729 |
| 140 | M | N | 0.00000 | 0.00000 | 0.0475000 | 0.000000 | 0.12500 | 0.12500 | 0.0609790 |
| 146 | N | P | 0.00000 | 0.00000 | 0.0237500 | 0.000000 | 0.06250 | 0.06250 | 0.0304895 |
| 147 | N | Q | 0.00000 | 0.00000 | 0.0475000 | 0.000000 | 0.12500 | 0.12500 | 0.0609790 |
A much larger simulation
set.seed(1L)
n <- 1000L
simKinships <- createSimKinships(ped, allSimParents, pop = ped$id, n = n)
kValues <- kinshipMatricesToKValues(simKinships)
extractKValue(kValues, id1 = "A", id2 = "F", simulation = 1L:10L)## [1] "sim_1" "sim_2" "sim_3" "sim_4" "sim_5" "sim_6" "sim_7" "sim_8"
## [9] "sim_9" "sim_10"
counts <- countKinshipValues(kValues)
counts$kinshipIds[1L:3L]## NULL
counts$kinshipValues[1L:3L]## NULL
counts$kinshipCounts[1L:3L]## NULL
stats_1000 <- summarizeKinshipValues(counts)
nrow(stats_1000[stats_1000$sd > 0.0, ])## [1] 94
kable(stats_1000[stats_1000$sd > 0.0, ], longtable = TRUE) %>%
kable_styling(
latex_options = c("striped", "repeat_header"),
repeat_header_method = "replace",
repeat_header_text = "\\textit{(continued)}"
)| id_1 | id_2 | min | secondQuartile | mean | median | thirdQuartile | max | sd | |
|---|---|---|---|---|---|---|---|---|---|
| 5 | A | E | 0.00000 | 0.00000 | 0.0792500 | 0.00000 | 0.25000 | 0.25000 | 0.1163851 |
| 6 | A | F | 0.12500 | 0.12500 | 0.1646250 | 0.12500 | 0.25000 | 0.25000 | 0.0581925 |
| 7 | A | G | 0.12500 | 0.12500 | 0.1646250 | 0.12500 | 0.25000 | 0.25000 | 0.0581925 |
| 9 | A | I | 0.25000 | 0.25000 | 0.2886250 | 0.25000 | 0.37500 | 0.37500 | 0.0577890 |
| 10 | A | J | 0.00000 | 0.00000 | 0.0772500 | 0.00000 | 0.25000 | 0.25000 | 0.1155781 |
| 11 | A | K | 0.00000 | 0.00000 | 0.0910000 | 0.00000 | 0.25000 | 0.25000 | 0.1203473 |
| 12 | A | L | 0.12500 | 0.12500 | 0.1705000 | 0.12500 | 0.25000 | 0.25000 | 0.0601737 |
| 14 | A | N | 0.00000 | 0.00000 | 0.0840000 | 0.00000 | 0.25000 | 0.25000 | 0.1181438 |
| 21 | B | E | 0.00000 | 0.00000 | 0.1200000 | 0.00000 | 0.25000 | 0.25000 | 0.1249625 |
| 22 | B | F | 0.12500 | 0.12500 | 0.1850000 | 0.12500 | 0.25000 | 0.25000 | 0.0624812 |
| 23 | B | G | 0.12500 | 0.12500 | 0.1850000 | 0.12500 | 0.25000 | 0.25000 | 0.0624812 |
| 25 | B | I | 0.00000 | 0.00000 | 0.0558750 | 0.00000 | 0.12500 | 0.12500 | 0.0621790 |
| 26 | B | J | 0.00000 | 0.00000 | 0.1117500 | 0.00000 | 0.25000 | 0.25000 | 0.1243580 |
| 30 | B | N | 0.00000 | 0.00000 | 0.1207500 | 0.00000 | 0.25000 | 0.25000 | 0.1249902 |
| 36 | C | E | 0.00000 | 0.00000 | 0.0996250 | 0.12500 | 0.12500 | 0.25000 | 0.0859018 |
| 37 | C | F | 0.12500 | 0.12500 | 0.1748125 | 0.18750 | 0.18750 | 0.25000 | 0.0429509 |
| 38 | C | G | 0.12500 | 0.12500 | 0.1748125 | 0.18750 | 0.18750 | 0.25000 | 0.0429509 |
| 40 | C | I | 0.12500 | 0.12500 | 0.1722500 | 0.18750 | 0.18750 | 0.25000 | 0.0411218 |
| 41 | C | J | 0.00000 | 0.00000 | 0.0945000 | 0.12500 | 0.12500 | 0.25000 | 0.0822436 |
| 42 | C | K | 0.00000 | 0.00000 | 0.0455000 | 0.00000 | 0.12500 | 0.12500 | 0.0601737 |
| 43 | C | L | 0.25000 | 0.25000 | 0.2727500 | 0.25000 | 0.31250 | 0.31250 | 0.0300868 |
| 45 | C | N | 0.00000 | 0.00000 | 0.1023750 | 0.12500 | 0.12500 | 0.25000 | 0.0855778 |
| 50 | D | E | 0.00000 | 0.00000 | 0.0996250 | 0.12500 | 0.12500 | 0.25000 | 0.0859018 |
| 51 | D | F | 0.25000 | 0.25000 | 0.2998125 | 0.31250 | 0.31250 | 0.37500 | 0.0429509 |
| 52 | D | G | 0.25000 | 0.25000 | 0.2998125 | 0.31250 | 0.31250 | 0.37500 | 0.0429509 |
| 54 | D | I | 0.12500 | 0.12500 | 0.1722500 | 0.18750 | 0.18750 | 0.25000 | 0.0411218 |
| 55 | D | J | 0.00000 | 0.00000 | 0.0945000 | 0.12500 | 0.12500 | 0.25000 | 0.0822436 |
| 56 | D | K | 0.00000 | 0.00000 | 0.0455000 | 0.00000 | 0.12500 | 0.12500 | 0.0601737 |
| 57 | D | L | 0.12500 | 0.12500 | 0.1477500 | 0.12500 | 0.18750 | 0.18750 | 0.0300868 |
| 59 | D | N | 0.00000 | 0.00000 | 0.1023750 | 0.12500 | 0.12500 | 0.25000 | 0.0855778 |
| 64 | E | F | 0.25000 | 0.25000 | 0.2998125 | 0.31250 | 0.31250 | 0.37500 | 0.0429509 |
| 65 | E | G | 0.25000 | 0.25000 | 0.2998125 | 0.31250 | 0.31250 | 0.37500 | 0.0429509 |
| 66 | E | H | 0.00000 | 0.00000 | 0.0996250 | 0.12500 | 0.12500 | 0.25000 | 0.0859018 |
| 67 | E | I | 0.00000 | 0.00000 | 0.0670000 | 0.06250 | 0.12500 | 0.25000 | 0.0720825 |
| 68 | E | J | 0.00000 | 0.00000 | 0.0547500 | 0.00000 | 0.12500 | 0.25000 | 0.0740045 |
| 69 | E | K | 0.00000 | 0.00000 | 0.0278750 | 0.00000 | 0.00000 | 0.12500 | 0.0520583 |
| 70 | E | L | 0.00000 | 0.00000 | 0.0637500 | 0.06250 | 0.12500 | 0.18750 | 0.0527491 |
| 71 | E | M | 0.00000 | 0.00000 | 0.0396250 | 0.00000 | 0.12500 | 0.12500 | 0.0581925 |
| 72 | E | N | 0.00000 | 0.00000 | 0.0576250 | 0.00000 | 0.12500 | 0.25000 | 0.0744599 |
| 74 | E | P | 0.00000 | 0.00000 | 0.0198125 | 0.00000 | 0.06250 | 0.06250 | 0.0290963 |
| 75 | E | Q | 0.00000 | 0.00000 | 0.0396250 | 0.00000 | 0.12500 | 0.12500 | 0.0581925 |
| 76 | F | F | 0.50000 | 0.50000 | 0.5498125 | 0.56250 | 0.56250 | 0.62500 | 0.0429509 |
| 77 | F | G | 0.25000 | 0.25000 | 0.2998125 | 0.31250 | 0.31250 | 0.37500 | 0.0429509 |
| 78 | F | H | 0.12500 | 0.12500 | 0.1748125 | 0.18750 | 0.18750 | 0.25000 | 0.0429509 |
| 79 | F | I | 0.06250 | 0.06250 | 0.1196250 | 0.12500 | 0.15625 | 0.25000 | 0.0447920 |
| 80 | F | J | 0.00000 | 0.00000 | 0.0746250 | 0.06250 | 0.12500 | 0.25000 | 0.0651156 |
| 81 | F | K | 0.00000 | 0.00000 | 0.0366875 | 0.00000 | 0.06250 | 0.12500 | 0.0419673 |
| 82 | F | L | 0.06250 | 0.06250 | 0.1057500 | 0.09375 | 0.12500 | 0.18750 | 0.0305468 |
| 83 | F | M | 0.06250 | 0.06250 | 0.0823125 | 0.06250 | 0.12500 | 0.12500 | 0.0290963 |
| 84 | F | N | 0.00000 | 0.00000 | 0.0800000 | 0.06250 | 0.12500 | 0.25000 | 0.0662241 |
| 86 | F | P | 0.03125 | 0.03125 | 0.0411562 | 0.03125 | 0.06250 | 0.06250 | 0.0145481 |
| 87 | F | Q | 0.06250 | 0.06250 | 0.0823125 | 0.06250 | 0.12500 | 0.12500 | 0.0290963 |
| 88 | G | G | 0.50000 | 0.50000 | 0.5498125 | 0.56250 | 0.56250 | 0.62500 | 0.0429509 |
| 89 | G | H | 0.12500 | 0.12500 | 0.1748125 | 0.18750 | 0.18750 | 0.25000 | 0.0429509 |
| 90 | G | I | 0.06250 | 0.06250 | 0.1196250 | 0.12500 | 0.15625 | 0.25000 | 0.0447920 |
| 91 | G | J | 0.00000 | 0.00000 | 0.0746250 | 0.06250 | 0.12500 | 0.25000 | 0.0651156 |
| 92 | G | K | 0.00000 | 0.00000 | 0.0366875 | 0.00000 | 0.06250 | 0.12500 | 0.0419673 |
| 93 | G | L | 0.06250 | 0.06250 | 0.1057500 | 0.09375 | 0.12500 | 0.18750 | 0.0305468 |
| 94 | G | M | 0.06250 | 0.06250 | 0.0823125 | 0.06250 | 0.12500 | 0.12500 | 0.0290963 |
| 95 | G | N | 0.00000 | 0.00000 | 0.0800000 | 0.06250 | 0.12500 | 0.25000 | 0.0662241 |
| 97 | G | P | 0.03125 | 0.03125 | 0.0411562 | 0.03125 | 0.06250 | 0.06250 | 0.0145481 |
| 98 | G | Q | 0.06250 | 0.06250 | 0.0823125 | 0.06250 | 0.12500 | 0.12500 | 0.0290963 |
| 100 | H | I | 0.12500 | 0.12500 | 0.1722500 | 0.18750 | 0.18750 | 0.25000 | 0.0411218 |
| 101 | H | J | 0.00000 | 0.00000 | 0.0945000 | 0.12500 | 0.12500 | 0.25000 | 0.0822436 |
| 102 | H | K | 0.00000 | 0.00000 | 0.0455000 | 0.00000 | 0.12500 | 0.12500 | 0.0601737 |
| 103 | H | L | 0.12500 | 0.12500 | 0.1477500 | 0.12500 | 0.18750 | 0.18750 | 0.0300868 |
| 105 | H | N | 0.00000 | 0.00000 | 0.1023750 | 0.12500 | 0.12500 | 0.25000 | 0.0855778 |
| 109 | I | I | 0.50000 | 0.50000 | 0.5386250 | 0.50000 | 0.62500 | 0.62500 | 0.0577890 |
| 110 | I | J | 0.25000 | 0.25000 | 0.2886250 | 0.25000 | 0.37500 | 0.37500 | 0.0577890 |
| 111 | I | K | 0.00000 | 0.00000 | 0.0585000 | 0.00000 | 0.12500 | 0.18750 | 0.0684968 |
| 112 | I | L | 0.06250 | 0.06250 | 0.1153750 | 0.09375 | 0.12500 | 0.21875 | 0.0408748 |
| 113 | I | M | 0.12500 | 0.12500 | 0.1443125 | 0.12500 | 0.18750 | 0.18750 | 0.0288945 |
| 114 | I | N | 0.00000 | 0.00000 | 0.0693125 | 0.06250 | 0.12500 | 0.25000 | 0.0739647 |
| 116 | I | P | 0.06250 | 0.06250 | 0.0721562 | 0.06250 | 0.09375 | 0.09375 | 0.0144473 |
| 117 | I | Q | 0.12500 | 0.12500 | 0.1443125 | 0.12500 | 0.18750 | 0.18750 | 0.0288945 |
| 119 | J | K | 0.00000 | 0.00000 | 0.0260000 | 0.00000 | 0.00000 | 0.12500 | 0.0507600 |
| 120 | J | L | 0.00000 | 0.00000 | 0.0602500 | 0.06250 | 0.06250 | 0.18750 | 0.0518182 |
| 121 | J | M | 0.00000 | 0.00000 | 0.0386250 | 0.00000 | 0.12500 | 0.12500 | 0.0577890 |
| 122 | J | N | 0.00000 | 0.00000 | 0.0546250 | 0.00000 | 0.12500 | 0.25000 | 0.0731409 |
| 124 | J | P | 0.00000 | 0.00000 | 0.0193125 | 0.00000 | 0.06250 | 0.06250 | 0.0288945 |
| 125 | J | Q | 0.00000 | 0.00000 | 0.0386250 | 0.00000 | 0.12500 | 0.12500 | 0.0577890 |
| 127 | K | L | 0.25000 | 0.25000 | 0.2727500 | 0.25000 | 0.31250 | 0.31250 | 0.0300868 |
| 128 | K | M | 0.00000 | 0.00000 | 0.0455000 | 0.00000 | 0.12500 | 0.12500 | 0.0601737 |
| 129 | K | N | 0.00000 | 0.00000 | 0.0285000 | 0.00000 | 0.00000 | 0.12500 | 0.0524691 |
| 131 | K | P | 0.00000 | 0.00000 | 0.0227500 | 0.00000 | 0.06250 | 0.06250 | 0.0300868 |
| 132 | K | Q | 0.00000 | 0.00000 | 0.0455000 | 0.00000 | 0.12500 | 0.12500 | 0.0601737 |
| 133 | L | L | 0.50000 | 0.50000 | 0.5227500 | 0.50000 | 0.56250 | 0.56250 | 0.0300868 |
| 134 | L | M | 0.06250 | 0.06250 | 0.0852500 | 0.06250 | 0.12500 | 0.12500 | 0.0300868 |
| 135 | L | N | 0.00000 | 0.00000 | 0.0654375 | 0.06250 | 0.12500 | 0.18750 | 0.0533825 |
| 137 | L | P | 0.03125 | 0.03125 | 0.0426250 | 0.03125 | 0.06250 | 0.06250 | 0.0150434 |
| 138 | L | Q | 0.06250 | 0.06250 | 0.0852500 | 0.06250 | 0.12500 | 0.12500 | 0.0300868 |
| 140 | M | N | 0.00000 | 0.00000 | 0.0420000 | 0.00000 | 0.12500 | 0.12500 | 0.0590719 |
| 146 | N | P | 0.00000 | 0.00000 | 0.0210000 | 0.00000 | 0.06250 | 0.06250 | 0.0295360 |
| 147 | N | Q | 0.00000 | 0.00000 | 0.0420000 | 0.00000 | 0.12500 | 0.12500 | 0.0590719 |
Comparing the values and variation found for the various kinship values:
stats_short <- stats_10[stats_10$sd > 0.0, ]
stats_long <- stats_1000[stats_1000$sd > 0.0, ]
if (any(stats_short$id_1 != stats_long$id_1) ||
any(stats_short$id_2 != stats_long$id_2)) {
cat("At least one row represents a different animal pair")
}
comprison <- data.frame(
id_1 = stats_short$id_1,
id_2 = stats_short$id_2,
meanKin_short = stats_short$mean,
meanKin_long = stats_long$mean,
meanKinDelta = abs(stats_short$mean - stats_long$mean),
sdKin_short = stats_short$sd,
sdKin_long = stats_long$sd,
sdKinDelta = abs(stats_short$sd - stats_long$sd)
)
kable(comprison,
longtable = TRUE,
digits = c(0L, 0L, 4L, 4L, 4L, 4L, 4L, 4L),
caption = stri_c(
"Comparision of estimated kinships between simulations ",
"of 10 (short) and 1000 (long)"
)
) %>%
kable_styling(
latex_options = c("striped", "repeat_header"),
repeat_header_method = "replace",
repeat_header_text = "\\textit{(continued)}",
font_size = 10L
)| id_1 | id_2 | meanKin_short | meanKin_long | meanKinDelta | sdKin_short | sdKin_long | sdKinDelta |
|---|---|---|---|---|---|---|---|
| A | E | 0.0750 | 0.0792 | 0.0043 | 0.1208 | 0.1164 | 0.0044 |
| A | F | 0.1625 | 0.1646 | 0.0021 | 0.0604 | 0.0582 | 0.0022 |
| A | G | 0.1625 | 0.1646 | 0.0021 | 0.0604 | 0.0582 | 0.0022 |
| A | I | 0.2625 | 0.2886 | 0.0261 | 0.0395 | 0.0578 | 0.0183 |
| A | J | 0.0250 | 0.0772 | 0.0522 | 0.0791 | 0.1156 | 0.0365 |
| A | K | 0.1250 | 0.0910 | 0.0340 | 0.1318 | 0.1203 | 0.0114 |
| A | L | 0.1875 | 0.1705 | 0.0170 | 0.0659 | 0.0602 | 0.0057 |
| A | N | 0.1000 | 0.0840 | 0.0160 | 0.1291 | 0.1181 | 0.0110 |
| B | E | 0.1500 | 0.1200 | 0.0300 | 0.1291 | 0.1250 | 0.0041 |
| B | F | 0.2000 | 0.1850 | 0.0150 | 0.0645 | 0.0625 | 0.0021 |
| B | G | 0.2000 | 0.1850 | 0.0150 | 0.0645 | 0.0625 | 0.0021 |
| B | I | 0.0625 | 0.0559 | 0.0066 | 0.0659 | 0.0622 | 0.0037 |
| B | J | 0.1250 | 0.1118 | 0.0132 | 0.1318 | 0.1244 | 0.0074 |
| B | N | 0.1000 | 0.1207 | 0.0207 | 0.1291 | 0.1250 | 0.0041 |
| C | E | 0.1125 | 0.0996 | 0.0129 | 0.1094 | 0.0859 | 0.0235 |
| C | F | 0.1812 | 0.1748 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| C | G | 0.1812 | 0.1748 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| C | I | 0.1625 | 0.1722 | 0.0097 | 0.0437 | 0.0411 | 0.0026 |
| C | J | 0.0750 | 0.0945 | 0.0195 | 0.0874 | 0.0822 | 0.0052 |
| C | K | 0.0625 | 0.0455 | 0.0170 | 0.0659 | 0.0602 | 0.0057 |
| C | L | 0.2812 | 0.2728 | 0.0085 | 0.0329 | 0.0301 | 0.0029 |
| C | N | 0.1000 | 0.1024 | 0.0024 | 0.0527 | 0.0856 | 0.0329 |
| D | E | 0.1125 | 0.0996 | 0.0129 | 0.1094 | 0.0859 | 0.0235 |
| D | F | 0.3062 | 0.2998 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| D | G | 0.3062 | 0.2998 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| D | I | 0.1625 | 0.1722 | 0.0097 | 0.0437 | 0.0411 | 0.0026 |
| D | J | 0.0750 | 0.0945 | 0.0195 | 0.0874 | 0.0822 | 0.0052 |
| D | K | 0.0625 | 0.0455 | 0.0170 | 0.0659 | 0.0602 | 0.0057 |
| D | L | 0.1562 | 0.1478 | 0.0085 | 0.0329 | 0.0301 | 0.0029 |
| D | N | 0.1000 | 0.1024 | 0.0024 | 0.0527 | 0.0856 | 0.0329 |
| E | F | 0.3062 | 0.2998 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| E | G | 0.3062 | 0.2998 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| E | H | 0.1125 | 0.0996 | 0.0129 | 0.1094 | 0.0859 | 0.0235 |
| E | I | 0.0688 | 0.0670 | 0.0018 | 0.0906 | 0.0721 | 0.0185 |
| E | J | 0.0625 | 0.0548 | 0.0078 | 0.0884 | 0.0740 | 0.0144 |
| E | K | 0.0875 | 0.0279 | 0.0596 | 0.0604 | 0.0521 | 0.0083 |
| E | L | 0.1000 | 0.0638 | 0.0363 | 0.0734 | 0.0527 | 0.0206 |
| E | M | 0.0375 | 0.0396 | 0.0021 | 0.0604 | 0.0582 | 0.0022 |
| E | N | 0.0500 | 0.0576 | 0.0076 | 0.0874 | 0.0745 | 0.0129 |
| E | P | 0.0187 | 0.0198 | 0.0011 | 0.0302 | 0.0291 | 0.0011 |
| E | Q | 0.0375 | 0.0396 | 0.0021 | 0.0604 | 0.0582 | 0.0022 |
| F | F | 0.5562 | 0.5498 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| F | G | 0.3062 | 0.2998 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| F | H | 0.1812 | 0.1748 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| F | I | 0.1156 | 0.1196 | 0.0040 | 0.0608 | 0.0448 | 0.0160 |
| F | J | 0.0688 | 0.0746 | 0.0059 | 0.0804 | 0.0651 | 0.0153 |
| F | K | 0.0750 | 0.0367 | 0.0383 | 0.0395 | 0.0420 | 0.0024 |
| F | L | 0.1281 | 0.1057 | 0.0224 | 0.0453 | 0.0305 | 0.0147 |
| F | M | 0.0813 | 0.0823 | 0.0011 | 0.0302 | 0.0291 | 0.0011 |
| F | N | 0.0750 | 0.0800 | 0.0050 | 0.0574 | 0.0662 | 0.0088 |
| F | P | 0.0406 | 0.0412 | 0.0005 | 0.0151 | 0.0145 | 0.0005 |
| F | Q | 0.0813 | 0.0823 | 0.0011 | 0.0302 | 0.0291 | 0.0011 |
| G | G | 0.5562 | 0.5498 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| G | H | 0.1812 | 0.1748 | 0.0064 | 0.0547 | 0.0430 | 0.0118 |
| G | I | 0.1156 | 0.1196 | 0.0040 | 0.0608 | 0.0448 | 0.0160 |
| G | J | 0.0688 | 0.0746 | 0.0059 | 0.0804 | 0.0651 | 0.0153 |
| G | K | 0.0750 | 0.0367 | 0.0383 | 0.0395 | 0.0420 | 0.0024 |
| G | L | 0.1281 | 0.1057 | 0.0224 | 0.0453 | 0.0305 | 0.0147 |
| G | M | 0.0813 | 0.0823 | 0.0011 | 0.0302 | 0.0291 | 0.0011 |
| G | N | 0.0750 | 0.0800 | 0.0050 | 0.0574 | 0.0662 | 0.0088 |
| G | P | 0.0406 | 0.0412 | 0.0005 | 0.0151 | 0.0145 | 0.0005 |
| G | Q | 0.0813 | 0.0823 | 0.0011 | 0.0302 | 0.0291 | 0.0011 |
| H | I | 0.1625 | 0.1722 | 0.0097 | 0.0437 | 0.0411 | 0.0026 |
| H | J | 0.0750 | 0.0945 | 0.0195 | 0.0874 | 0.0822 | 0.0052 |
| H | K | 0.0625 | 0.0455 | 0.0170 | 0.0659 | 0.0602 | 0.0057 |
| H | L | 0.1562 | 0.1478 | 0.0085 | 0.0329 | 0.0301 | 0.0029 |
| H | N | 0.1000 | 0.1024 | 0.0024 | 0.0527 | 0.0856 | 0.0329 |
| I | I | 0.5125 | 0.5386 | 0.0261 | 0.0395 | 0.0578 | 0.0183 |
| I | J | 0.2625 | 0.2886 | 0.0261 | 0.0395 | 0.0578 | 0.0183 |
| I | K | 0.0750 | 0.0585 | 0.0165 | 0.0710 | 0.0685 | 0.0025 |
| I | L | 0.1187 | 0.1154 | 0.0034 | 0.0506 | 0.0409 | 0.0097 |
| I | M | 0.1312 | 0.1443 | 0.0131 | 0.0198 | 0.0289 | 0.0091 |
| I | N | 0.0688 | 0.0693 | 0.0006 | 0.0688 | 0.0740 | 0.0052 |
| I | P | 0.0656 | 0.0722 | 0.0065 | 0.0099 | 0.0144 | 0.0046 |
| I | Q | 0.1312 | 0.1443 | 0.0131 | 0.0198 | 0.0289 | 0.0091 |
| J | K | 0.0250 | 0.0260 | 0.0010 | 0.0527 | 0.0508 | 0.0019 |
| J | L | 0.0500 | 0.0602 | 0.0102 | 0.0645 | 0.0518 | 0.0127 |
| J | M | 0.0125 | 0.0386 | 0.0261 | 0.0395 | 0.0578 | 0.0183 |
| J | N | 0.0375 | 0.0546 | 0.0171 | 0.0604 | 0.0731 | 0.0128 |
| J | P | 0.0063 | 0.0193 | 0.0131 | 0.0198 | 0.0289 | 0.0091 |
| J | Q | 0.0125 | 0.0386 | 0.0261 | 0.0395 | 0.0578 | 0.0183 |
| K | L | 0.2812 | 0.2728 | 0.0085 | 0.0329 | 0.0301 | 0.0029 |
| K | M | 0.0625 | 0.0455 | 0.0170 | 0.0659 | 0.0602 | 0.0057 |
| K | N | 0.0500 | 0.0285 | 0.0215 | 0.0645 | 0.0525 | 0.0121 |
| K | P | 0.0312 | 0.0227 | 0.0085 | 0.0329 | 0.0301 | 0.0029 |
| K | Q | 0.0625 | 0.0455 | 0.0170 | 0.0659 | 0.0602 | 0.0057 |
| L | L | 0.5312 | 0.5228 | 0.0085 | 0.0329 | 0.0301 | 0.0029 |
| L | M | 0.0938 | 0.0853 | 0.0085 | 0.0329 | 0.0301 | 0.0029 |
| L | N | 0.0750 | 0.0654 | 0.0096 | 0.0493 | 0.0534 | 0.0041 |
| L | P | 0.0469 | 0.0426 | 0.0042 | 0.0165 | 0.0150 | 0.0014 |
| L | Q | 0.0938 | 0.0853 | 0.0085 | 0.0329 | 0.0301 | 0.0029 |
| M | N | 0.0500 | 0.0420 | 0.0080 | 0.0645 | 0.0591 | 0.0055 |
| N | P | 0.0250 | 0.0210 | 0.0040 | 0.0323 | 0.0295 | 0.0027 |
| N | Q | 0.0500 | 0.0420 | 0.0080 | 0.0645 | 0.0591 | 0.0055 |
stats_short <- stats_100[stats_100$sd > 0.0, ]
stats_long <- stats_1000[stats_1000$sd > 0.0, ]
if (any(stats_short$id_1 != stats_long$id_1) ||
any(stats_short$id_2 != stats_long$id_2)) {
cat("At least one row represents a different animal pair")
}
comprison <- data.frame(
id_1 = stats_short$id_1,
id_2 = stats_short$id_2,
meanKin_short = stats_short$mean,
meanKin_long = stats_long$mean,
meanKinDelta = abs(stats_short$mean - stats_long$mean),
sdKin_short = stats_short$sd,
sdKin_long = stats_long$sd,
sdKinDelta = abs(stats_short$sd - stats_long$sd)
)
kable(comprison,
longtable = TRUE,
digits = c(0L, 0L, 4L, 4L, 4L, 4L, 4L, 4L),
caption = stri_c(
"Comparision of estimated kinships between simulations ",
"of 100 (short) and 1000 (long)"
)
) %>%
kable_styling(
latex_options = c("striped", "repeat_header"),
repeat_header_method = "replace",
repeat_header_text = "\\textit{(continued)}",
font_size = 10L
)| id_1 | id_2 | meanKin_short | meanKin_long | meanKinDelta | sdKin_short | sdKin_long | sdKinDelta |
|---|---|---|---|---|---|---|---|
| A | E | 0.0725 | 0.0792 | 0.0068 | 0.1140 | 0.1164 | 0.0024 |
| A | F | 0.1612 | 0.1646 | 0.0034 | 0.0570 | 0.0582 | 0.0012 |
| A | G | 0.1612 | 0.1646 | 0.0034 | 0.0570 | 0.0582 | 0.0012 |
| A | I | 0.2825 | 0.2886 | 0.0061 | 0.0551 | 0.0578 | 0.0027 |
| A | J | 0.0650 | 0.0772 | 0.0122 | 0.1102 | 0.1156 | 0.0054 |
| A | K | 0.1000 | 0.0910 | 0.0090 | 0.1231 | 0.1203 | 0.0027 |
| A | L | 0.1750 | 0.1705 | 0.0045 | 0.0615 | 0.0602 | 0.0014 |
| A | N | 0.0950 | 0.0840 | 0.0110 | 0.1220 | 0.1181 | 0.0038 |
| B | E | 0.1225 | 0.1200 | 0.0025 | 0.1256 | 0.1250 | 0.0006 |
| B | F | 0.1862 | 0.1850 | 0.0013 | 0.0628 | 0.0625 | 0.0003 |
| B | G | 0.1862 | 0.1850 | 0.0013 | 0.0628 | 0.0625 | 0.0003 |
| B | I | 0.0512 | 0.0559 | 0.0046 | 0.0618 | 0.0622 | 0.0004 |
| B | J | 0.1025 | 0.1118 | 0.0093 | 0.1236 | 0.1244 | 0.0008 |
| B | N | 0.1325 | 0.1207 | 0.0118 | 0.1254 | 0.1250 | 0.0004 |
| C | E | 0.0975 | 0.0996 | 0.0021 | 0.0863 | 0.0859 | 0.0004 |
| C | F | 0.1737 | 0.1748 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| C | G | 0.1737 | 0.1748 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| C | I | 0.1669 | 0.1722 | 0.0054 | 0.0377 | 0.0411 | 0.0034 |
| C | J | 0.0838 | 0.0945 | 0.0107 | 0.0755 | 0.0822 | 0.0068 |
| C | K | 0.0500 | 0.0455 | 0.0045 | 0.0615 | 0.0602 | 0.0014 |
| C | L | 0.2750 | 0.2728 | 0.0023 | 0.0308 | 0.0301 | 0.0007 |
| C | N | 0.1138 | 0.1024 | 0.0114 | 0.0890 | 0.0856 | 0.0034 |
| D | E | 0.0975 | 0.0996 | 0.0021 | 0.0863 | 0.0859 | 0.0004 |
| D | F | 0.2988 | 0.2998 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| D | G | 0.2988 | 0.2998 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| D | I | 0.1669 | 0.1722 | 0.0054 | 0.0377 | 0.0411 | 0.0034 |
| D | J | 0.0838 | 0.0945 | 0.0107 | 0.0755 | 0.0822 | 0.0068 |
| D | K | 0.0500 | 0.0455 | 0.0045 | 0.0615 | 0.0602 | 0.0014 |
| D | L | 0.1500 | 0.1478 | 0.0023 | 0.0308 | 0.0301 | 0.0007 |
| D | N | 0.1138 | 0.1024 | 0.0114 | 0.0890 | 0.0856 | 0.0034 |
| E | F | 0.2988 | 0.2998 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| E | G | 0.2988 | 0.2998 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| E | H | 0.0975 | 0.0996 | 0.0021 | 0.0863 | 0.0859 | 0.0004 |
| E | I | 0.0644 | 0.0670 | 0.0026 | 0.0702 | 0.0721 | 0.0019 |
| E | J | 0.0562 | 0.0548 | 0.0015 | 0.0782 | 0.0740 | 0.0042 |
| E | K | 0.0312 | 0.0279 | 0.0034 | 0.0544 | 0.0521 | 0.0023 |
| E | L | 0.0644 | 0.0638 | 0.0006 | 0.0579 | 0.0527 | 0.0051 |
| E | M | 0.0362 | 0.0396 | 0.0034 | 0.0570 | 0.0582 | 0.0012 |
| E | N | 0.0587 | 0.0576 | 0.0011 | 0.0742 | 0.0745 | 0.0002 |
| E | P | 0.0181 | 0.0198 | 0.0017 | 0.0285 | 0.0291 | 0.0006 |
| E | Q | 0.0362 | 0.0396 | 0.0034 | 0.0570 | 0.0582 | 0.0012 |
| F | F | 0.5488 | 0.5498 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| F | G | 0.2988 | 0.2998 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| F | H | 0.1737 | 0.1748 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| F | I | 0.1156 | 0.1196 | 0.0040 | 0.0429 | 0.0448 | 0.0018 |
| F | J | 0.0700 | 0.0746 | 0.0046 | 0.0624 | 0.0651 | 0.0028 |
| F | K | 0.0406 | 0.0367 | 0.0039 | 0.0439 | 0.0420 | 0.0019 |
| F | L | 0.1072 | 0.1057 | 0.0014 | 0.0342 | 0.0305 | 0.0036 |
| F | M | 0.0806 | 0.0823 | 0.0017 | 0.0285 | 0.0291 | 0.0006 |
| F | N | 0.0862 | 0.0800 | 0.0062 | 0.0681 | 0.0662 | 0.0019 |
| F | P | 0.0403 | 0.0412 | 0.0008 | 0.0143 | 0.0145 | 0.0003 |
| F | Q | 0.0806 | 0.0823 | 0.0017 | 0.0285 | 0.0291 | 0.0006 |
| G | G | 0.5488 | 0.5498 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| G | H | 0.1737 | 0.1748 | 0.0011 | 0.0431 | 0.0430 | 0.0002 |
| G | I | 0.1156 | 0.1196 | 0.0040 | 0.0429 | 0.0448 | 0.0018 |
| G | J | 0.0700 | 0.0746 | 0.0046 | 0.0624 | 0.0651 | 0.0028 |
| G | K | 0.0406 | 0.0367 | 0.0039 | 0.0439 | 0.0420 | 0.0019 |
| G | L | 0.1072 | 0.1057 | 0.0014 | 0.0342 | 0.0305 | 0.0036 |
| G | M | 0.0806 | 0.0823 | 0.0017 | 0.0285 | 0.0291 | 0.0006 |
| G | N | 0.0862 | 0.0800 | 0.0062 | 0.0681 | 0.0662 | 0.0019 |
| G | P | 0.0403 | 0.0412 | 0.0008 | 0.0143 | 0.0145 | 0.0003 |
| G | Q | 0.0806 | 0.0823 | 0.0017 | 0.0285 | 0.0291 | 0.0006 |
| H | I | 0.1669 | 0.1722 | 0.0054 | 0.0377 | 0.0411 | 0.0034 |
| H | J | 0.0838 | 0.0945 | 0.0107 | 0.0755 | 0.0822 | 0.0068 |
| H | K | 0.0500 | 0.0455 | 0.0045 | 0.0615 | 0.0602 | 0.0014 |
| H | L | 0.1500 | 0.1478 | 0.0023 | 0.0308 | 0.0301 | 0.0007 |
| H | N | 0.1138 | 0.1024 | 0.0114 | 0.0890 | 0.0856 | 0.0034 |
| I | I | 0.5325 | 0.5386 | 0.0061 | 0.0551 | 0.0578 | 0.0027 |
| I | J | 0.2825 | 0.2886 | 0.0061 | 0.0551 | 0.0578 | 0.0027 |
| I | K | 0.0594 | 0.0585 | 0.0009 | 0.0655 | 0.0685 | 0.0030 |
| I | L | 0.1131 | 0.1154 | 0.0023 | 0.0382 | 0.0409 | 0.0027 |
| I | M | 0.1412 | 0.1443 | 0.0031 | 0.0276 | 0.0289 | 0.0013 |
| I | N | 0.0756 | 0.0693 | 0.0063 | 0.0761 | 0.0740 | 0.0021 |
| I | P | 0.0706 | 0.0722 | 0.0015 | 0.0138 | 0.0144 | 0.0007 |
| I | Q | 0.1412 | 0.1443 | 0.0031 | 0.0276 | 0.0289 | 0.0013 |
| J | K | 0.0187 | 0.0260 | 0.0072 | 0.0449 | 0.0508 | 0.0059 |
| J | L | 0.0512 | 0.0602 | 0.0090 | 0.0465 | 0.0518 | 0.0053 |
| J | M | 0.0325 | 0.0386 | 0.0061 | 0.0551 | 0.0578 | 0.0027 |
| J | N | 0.0562 | 0.0546 | 0.0016 | 0.0674 | 0.0731 | 0.0058 |
| J | P | 0.0163 | 0.0193 | 0.0031 | 0.0276 | 0.0289 | 0.0013 |
| J | Q | 0.0325 | 0.0386 | 0.0061 | 0.0551 | 0.0578 | 0.0027 |
| K | L | 0.2750 | 0.2728 | 0.0023 | 0.0308 | 0.0301 | 0.0007 |
| K | M | 0.0500 | 0.0455 | 0.0045 | 0.0615 | 0.0602 | 0.0014 |
| K | N | 0.0338 | 0.0285 | 0.0053 | 0.0558 | 0.0525 | 0.0033 |
| K | P | 0.0250 | 0.0227 | 0.0023 | 0.0308 | 0.0301 | 0.0007 |
| K | Q | 0.0500 | 0.0455 | 0.0045 | 0.0615 | 0.0602 | 0.0014 |
| L | L | 0.5250 | 0.5228 | 0.0022 | 0.0308 | 0.0301 | 0.0007 |
| L | M | 0.0875 | 0.0853 | 0.0022 | 0.0308 | 0.0301 | 0.0007 |
| L | N | 0.0737 | 0.0654 | 0.0083 | 0.0585 | 0.0534 | 0.0051 |
| L | P | 0.0437 | 0.0426 | 0.0011 | 0.0154 | 0.0150 | 0.0003 |
| L | Q | 0.0875 | 0.0853 | 0.0022 | 0.0308 | 0.0301 | 0.0007 |
| M | N | 0.0475 | 0.0420 | 0.0055 | 0.0610 | 0.0591 | 0.0019 |
| N | P | 0.0238 | 0.0210 | 0.0027 | 0.0305 | 0.0295 | 0.0010 |
| N | Q | 0.0475 | 0.0420 | 0.0055 | 0.0610 | 0.0591 | 0.0019 |